近日,我院生物治疗研究中心杨胜勇教授团队在自然杂志子刊Nature Communications发文,揭示了基于深度学习的分子生成模型发现RIPK1小分子抑制剂的研究过程,显示了生成式深度学习(generative deep learning, GDL)模型生成全新分子结构的能力,彰显了深度学习在药物发现方面的巨大潜力。四川大学生物治疗国家重点实验室李越山博士后、张丽婷博士、汪益妃博士和邹俊副研究员为论文共同第一作者,杨胜勇教授为论文通讯作者。

在药物研发初期,如何高效发现具有新型骨架结构的苗头化合物或先导化合物是创新药物研发的关键一步,同时也是一项极富挑战性的任务。传统的策略是利用高通量筛选方法从现有化合物库中进行筛选。然而,由于现有化合物库的结构多样性有限,以及各大制药公司和药物研发机构的反复筛选,发现具有自主知识产权的全新骨架结构的活性化合物已变得越来越困难。而基于深度学习的生成模型可以产生具有全新骨架结构的化合物,为解决传统高通量筛选方法遇到的困境提供了新思路。
近年来,基于深度学习的生成模型或生成式深度学习(generative deep learning, GDL)模型的研究获得了快速发展。其中,基于循环神经网络(recurrent neural network, RNN)的GDL模型研究报道最为广泛。而条件循环神经网络(conditional RNN, cRNN)通过给定RNN初始状态向量作为条件,可显式指导后续分子生成过程。然而,现有cRNN以及其他GDL模型仍然存在诸多缺陷,例如过度依赖目标函数,生成分子新颖性、多样性有限等。此外,尽管大多数GDL模型已在理论层面得到验证,但应用于实际创新药物发现并获得成功的例子仍很少。
为此,杨胜勇教授团队提出了一种新型cRNN分子生成模型,该模型整合了迁移学习(transfer learning)、正则化增强(regularization enhancement)和采样增强(sampling enhancement)等策略。团队进一步将该模型应用于RIPK1激酶抑制剂的发现。通过建立定制化的RIPK1抑制剂虚拟化合物库、虚拟筛选、化学合成和生物活性验证,在未经任何改构的情况下,即获得了一个高活性和选择性的RIPK1抑制剂(RI-962)。团队随后解析了RIPK1–RI-962的晶体结构,从结构上阐明了RI-962具有高活性和选择性的分子机制。团队还在TNFα诱导的全身炎症反应综合征(systemic inflammatory response syndrome, SIRS)和DSS诱导的炎症性肠病(inflammatory bowel disease, IBD)的小鼠模型上评价了RI-962的体内效果,结果显示RI-962通过抑制RIPK1激酶活性从而改善TNFα诱导SIRS和DSS诱导的IBD损伤。
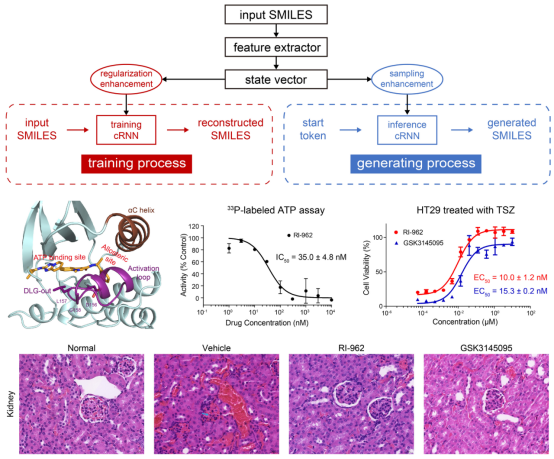
基于此,该研究团队提出了一种新的GDL模型,并利用该模型发现了一个高活性、选择性RIPK1抑制剂。该研究显示了GDL模型生成全新分子结构的能力,彰显了深度学习在药物发现方面的巨大潜力。
该研究工作得到了国家自然科学基金创新研究群体项目、重点项目、面上项目、青年科学基金项目,华西医院1·3·5工程和国家博士后创新人才计划等项目的大力支持。